上海科技大學寒假放假時間安排校歷及什么時候開學時間規定
更新:2023-09-21 23:26:02 高考升學網一、上海科技大學寒假放假時間及開學時間
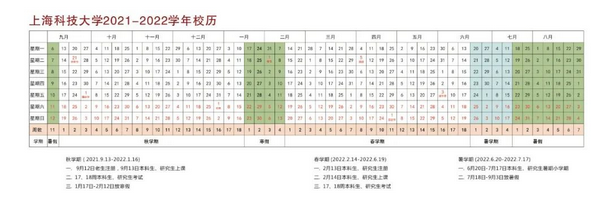

二、上海科技大學最新消息資訊
日,我校iHuman研究所水雯箐課題組與信息學院何旭明課題組合作在Nature Communications期刊發表了題為 “DeepPhospho accelerates DIA phosphoproteome profiling throughin silicolibrary generation” 的研究論文,探索利用新型深度神經網絡挖掘蛋白質組數據。該工作創建了DeepPhospho譜圖預測模型,并為數據非依賴型(DIA)磷酸化蛋白質組的數據解析提供了一套新流程。該流程能夠大幅提升對細胞內磷酸化蛋白質及其修飾位點的鑒定數目,同時保證位點鑒定和磷酸化水定量的高準確度和可重復性。利用該流程對同一套組學數據進行重新挖掘,相比于常規的生物信息學流程能夠發現更多的細胞生長因子介導的信號通路和調控的下游激酶。
蛋白質磷酸化是最常見和功能最重要的一種翻譯后修飾,幾乎所有的細胞信號通路都受到磷酸化修飾的精密調控。年來,基于質譜的磷酸化蛋白質組研究描繪出大量蛋白質的磷酸化修飾圖譜,極大程度地加深了對信號轉導網絡調控的全局性認識,也發現了不少與信號通路失調相關的潛在藥物靶點。由于功能性磷酸化位點的修飾水通常較低,如何在高通量分析磷酸化修飾的同時保證位點鑒定的準確性,并獲得精確的定量調控信息,這是磷酸化蛋白質組學面臨的關鍵技術挑戰。
數據非依賴性(DIA)數據采集是一種新型的質譜數據采集方法,理論上可以數字化保存生物樣品蛋白質組的全部信息。但DIA數據結構極為復雜,數據解析通常需要在預實驗中建立大容量的參考譜圖庫,這個建立參考庫的要求明顯加大了DIA組學實驗的難度和樣品消耗量。本研究工作首先建立了DeepPhospho神經網絡模型,用于對DIA譜圖數據的預測(圖1),并在性能測試上優于已發表的譜圖預測模型。而后,研究者利用DeepPhospho構建完整的人源磷酸化蛋白質組的預測譜圖庫(圖2),借助迭代式檢索策略對DIA質譜數據進行深度挖掘。對同一套數據的比較研究發現,該新流程能獲得數目最多的磷酸化肽段與磷酸化位點的序列和定量信息,并且省去對實驗參考庫的需求,顯著簡化了實驗過程。為便于新工具的推廣使用,DeepPhospho還提供了在線網站版和離線工具包。
上海科技大學水雯箐教授和何旭明教授為該工作的共同通訊作者,生命學院博士研究生婁容琿和信息學院碩士研究生劉偉振為共同第一作者,信息學院博士研究生李榮頡和iHuman研究所研究助理李珊珊為本課題做出了重要貢獻。上海科技大學為第一完成單位。該工作得到了科技部、國家自然科學基金、上海市科委以及上科大科研啟動基金的支持。
三、上海科技大學相關文章分享
上海師范大學天華學院寒假放假時間安排校歷及什么時候開學時間規定2023-09-21 11:42:25
上海外國語大學賢達經濟人文學院寒假放假時間安排校歷及什么時候開2023-09-21 02:13:01
2025年上海科技大學轉專業的要求條件,轉專業難不難好轉嗎 2023-09-19 04:28:12
2025年上海科技大學新生宿舍條件圖片環境怎么樣,有獨立衛生間嗎 2023-09-15 17:42:22
2025年上海科技大學錄取通知書什么時候發放,發放時間及查詢網址入口 2023-09-14 19:25:15
2025年上海科技大學錄取結果查詢什么時候公布 附查詢入口時間 2023-09-16 22:33:34
上海科技大學開設專業有哪些 附專業介紹2023-09-16 22:56:38
上海科技大學是985大學嗎2023-09-19 11:02:36
上海科技大學是211大學嗎2023-09-17 06:02:29
上海科技大學是幾本大學2023-09-21 14:30:43
上海科技大學是一本大學嗎2023-09-14 13:59:08
上海科技大學有幾個校區,哪個校區最好及各校區介紹 2023-09-16 13:54:27
東北大學秦皇島分校和河北工業大學哪個好 分數線排名對比2025-05-23 17:06:03
江西中醫藥大學和淮陰工學院哪個好 分數線排名對比2025-05-23 17:04:53
天津外國語大學和山西財經大學哪個好 分數線排名對比2025-05-23 17:03:25
蘭州交通大學在廣東預估錄取分數線多少分2025-05-23 17:01:58
石河子大學和渤海大學哪個好 分數線排名對比2025-05-23 17:00:42
青海高考理科類529分能上什么大學(公辦+民辦)2025-05-23 16:59:30
廣州南洋理工職業學院四川錄取分數線及招生人數 附-2020最低位次排名2025-05-23 16:58:01
浙江高考排名在236950的考生能報什么大學(原創)2025-05-23 16:56:35
三亞中瑞酒店管理職業學院和江蘇財會職業學院哪個好 分數線排名對比2025-05-23 16:55:20
曲靖職業技術學院的旅游管理專業分數線(附2020-最低分排名怎么樣)2025-05-23 16:54:11
長安大學的材料成型及控制工程專業分數線(附2020-最低分排名怎么樣)2025-05-23 16:52:56
吉林電子信息職業技術學院在甘肅錄取分數線是多少?最低位次排名2025-05-23 16:51:41
上海師范大學天華學院寒假放假時間安排校歷及什么時候開學時間規定2023-09-21 11:42:25
上海外國語大學賢達經濟人文學院寒假放假時間安排校歷及什么時候開2023-09-21 02:13:01
 上海師范大學天華學院寒假放假時間安排校歷及什么時候開學時間規定
上海師范大學天華學院寒假放假時間安排校歷及什么時候開學時間規定 上海外國語大學賢達經濟人文學院寒假放假時間安排校歷及什么時候開
上海外國語大學賢達經濟人文學院寒假放假時間安排校歷及什么時候開 2025年上海科技大學轉專業的要求條件,轉專業難不難好轉嗎
2025年上海科技大學轉專業的要求條件,轉專業難不難好轉嗎  2025年上海科技大學新生宿舍條件圖片環境怎么樣,有獨立衛生間嗎
2025年上海科技大學新生宿舍條件圖片環境怎么樣,有獨立衛生間嗎  2025年上海科技大學錄取通知書什么時候發放,發放時間及查詢網址入口
2025年上海科技大學錄取通知書什么時候發放,發放時間及查詢網址入口  2025年上海科技大學錄取結果查詢什么時候公布 附查詢入口時間
2025年上海科技大學錄取結果查詢什么時候公布 附查詢入口時間  上海科技大學開設專業有哪些 附專業介紹
上海科技大學開設專業有哪些 附專業介紹 上海科技大學是985大學嗎
上海科技大學是985大學嗎 上海科技大學是211大學嗎
上海科技大學是211大學嗎 上海科技大學是幾本大學
上海科技大學是幾本大學 上海科技大學是一本大學嗎
上海科技大學是一本大學嗎 上海科技大學有幾個校區,哪個校區最好及各校區介紹
上海科技大學有幾個校區,哪個校區最好及各校區介紹  東北大學秦皇島分校和河北工業大學哪個好 分數線排名對比
東北大學秦皇島分校和河北工業大學哪個好 分數線排名對比 江西中醫藥大學和淮陰工學院哪個好 分數線排名對比
江西中醫藥大學和淮陰工學院哪個好 分數線排名對比 天津外國語大學和山西財經大學哪個好 分數線排名對比
天津外國語大學和山西財經大學哪個好 分數線排名對比 蘭州交通大學在廣東預估錄取分數線多少分
蘭州交通大學在廣東預估錄取分數線多少分 石河子大學和渤海大學哪個好 分數線排名對比
石河子大學和渤海大學哪個好 分數線排名對比 青海高考理科類529分能上什么大學(公辦+民辦)
青海高考理科類529分能上什么大學(公辦+民辦) 廣州南洋理工職業學院四川錄取分數線及招生人數 附-2020最低位次排名
廣州南洋理工職業學院四川錄取分數線及招生人數 附-2020最低位次排名 浙江高考排名在236950的考生能報什么大學(原創)
浙江高考排名在236950的考生能報什么大學(原創) 三亞中瑞酒店管理職業學院和江蘇財會職業學院哪個好 分數線排名對比
三亞中瑞酒店管理職業學院和江蘇財會職業學院哪個好 分數線排名對比 曲靖職業技術學院的旅游管理專業分數線(附2020-最低分排名怎么樣)
曲靖職業技術學院的旅游管理專業分數線(附2020-最低分排名怎么樣) 長安大學的材料成型及控制工程專業分數線(附2020-最低分排名怎么樣)
長安大學的材料成型及控制工程專業分數線(附2020-最低分排名怎么樣) 吉林電子信息職業技術學院在甘肅錄取分數線是多少?最低位次排名
吉林電子信息職業技術學院在甘肅錄取分數線是多少?最低位次排名



